Nyupptäckta gener för antibiotikaresistens kan leda till nya mediciner
Antibiotikaresistens76 nya gener som ger antibiotikaresistens har upptäckts genom analys av stora mängder DNA-sekvenser från olika bakterier. Flera av dessa hittills okända resistensgener gör att bakterier kan stå emot även den mest kraftfulla antibiotikan, visar forskare vid Chalmers och Göteborgs universitet. Forskningen öppnar upp för insatser mot spridning av nya former av multiresistenta bakterier.
Bakterier som inte längre går att behandla med antibiotika är ett snabbt växande globalt problem. Antibiotikaresistens i sjukdomsframkallande bakterier utvecklas inte bara genom mutationer utan också genom att de tar upp gener från andra, ofarliga bakterier.
Genom att analysera mycket stora mängder DNA lyckades forskarteamet från Göteborg beskriva 76 nya typer av resistensgener. Flera av dessa ger bakterierna förmågan att bryta ner karbapenemer, den allra kraftfullaste typen av antibiotika som idag ofta används för att behandla multiresistenta bakterier där inga andra antibiotika biter. Upptäckten har publicerats i den vetenskapliga tidskriften Microbiome.
– Vår studie visar att det finns väldigt många okända resistensgener. Kunskap om dessa gener gör det möjligt att tidigt hitta och förhoppningsvis hantera nya former av multiresistenta bakterier, säger Erik Kristiansson, professor i biostatistik och ledare för forskargruppen på Chalmers.
– Ju mer vi vet om hur bakterierna kan försvara sig mot antibiotika, desto större förutsättningar har vi att utveckla läkemedel som också står emot deras försvar, förklarar medförfattare Joakim Larsson, professor i miljöfarmakologi vid Göteborgs universitet.
Förutspår resistens
Resultaten i studien kommer från analys av DNA-sekvenser från bakterier insamlade från människor och olika miljöer runt om i världen.
– Resistensgener är ofta mycket ovanliga så stora mängder sekvensdata måste undersökas innan en ny gen kan påträffas, fortsätter Erik Kristiansson.
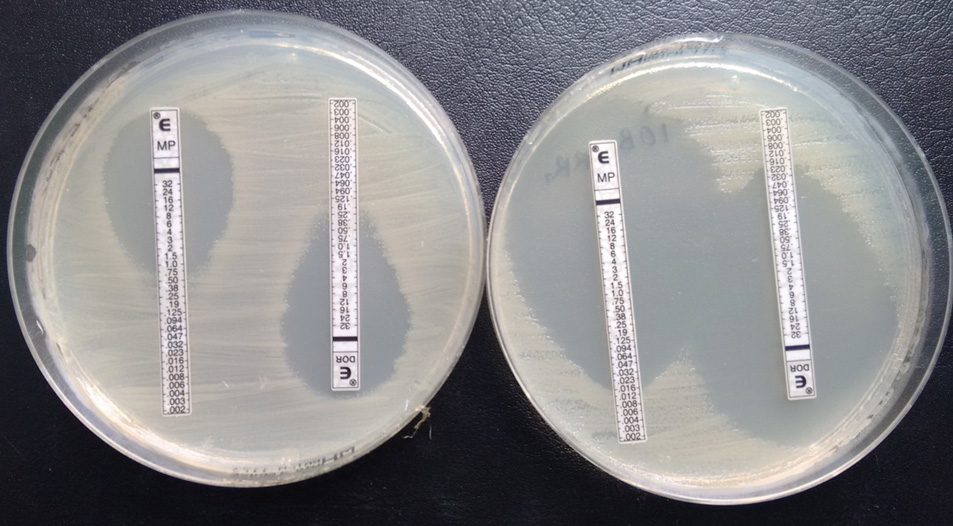
Det är dessutom svårt att veta vad som är en resistensgen om den inte tidigare är beskriven. Forskargruppen löste detta genom att utveckla nya beräkningsmetoder som letar efter speciella mönster i DNA kopplade till antibiotikaresistens. Genom att sedan testa de nya generna på labbet kunde forskarna visa att deras prediktioner var riktiga.
– För att hitta sådana mönster i DNA så använder vi metoder som är utvecklade för att hantera mycket stora datamängder, säger Fanny Berglund, doktorand i forskargruppen.
Nästa steg i forskningen är att söka efter gener som ger upphov till resistens mot ytterligare former av antibiotika.
– Våra resultat är bara toppen av ett isberg. Det finns en stor mängd resistensgener som vi idag inte har någon kännedom om och som kan komma att utgöra stora problem i framtiden, säger Erik Kristiansson.
För mer information:
Erik Kristiansson, Chalmers, 070-525 97 51, erik.kristiansson@chalmers.se