Internationella forskare studerar tre olika proteiner från coronaviruset SARS CoV-2 med hjälp av strålröret BioMAX vid MAX IV-laboratoriet i Lund. Nyligen lyckades de bestämma strukturen hos, och påbörja analysen av ett av dessa proteiner, Nsp10.
För att hitta ett effektivt läkemedel som kan stoppa det nya coronaviruset från att orsaka sjukdomen Covid-19 behöver forskarna förstå hur virusets förmåga att föröka sig kan blockeras. Viruset kommer då att ”dö ut” eftersom det misslyckas med att kopiera sin arvsmassa (replikera sitt RNA) inne i värdcellen och därefter göra infektiösa partiklar. Ett sätt att få denna kunskap är att använda intensiv röntgenstrålning som ger en detaljerad bild av strukturen hos de proteiner som är involverade i replikeringsprocessen.
16 proteiner för förökning
Det finns 16 så kallade icke-strukturella proteiner (Nsp:er) i SARS CoV-2, vilka spelar en viktig roll i den virala replikationen och transkriptionen, de första stegen i virusets förökning. Att störa virusets förmåga att replikera sig, och därmed kunna fortsätta att producera ett livskraftigt virus, är i fokus för många internationella forskare. Högupplösta kristallografiska och andra strukturbiologiska undersökningar spelar en viktig roll för att antingen hitta nya virushämmare eller för att studera hur befintliga läkemedel kan användas för att blockera det nya coronaviruset.
Replikation: Förökning av arvsmassan genom kopiering av DNA eller RNA inuti cellkärnan. Virus, som inte har ett eget replikationssystem, snyltar på värdcellens maskineri.
Transkripition: Översättning till proteiner, efter receptet i arvsmassan (DNA- eller RNA-koden). Virus, som inte har något eget transkriptionssystem, använder även här värdcellens maskineri. Proteinerna är nödvändiga för att bygga nya virus-molekyler.
Det finns flera proteiner involverade i kontrollmekanismen som viruset har för att skydda sin arvsmassa.
Ett internationellt samarbete mellan UCL School of Pharmacy, Lund Protein Production Platform (LP3) och ESS DEMAX Laboratory fokuserar på tre av dessa: Nsp10, Nsp14 och Nsp16, i syfte att identifiera små kemiska molekyler som kan ändra dessa proteiners funktioner.
Proteinkristaller led i utveckling av läkemedel
En viktig metod i läkemedelsutveckling heter Fragment-based ligand discovery (FBLD). Strategin med denna teknik är att man vill hitta flera små molekyler, fraktioner av ett möjligt komplett läkemedel, som binder eller hindrar proteinet man studerar. Dessa kan sedan sättas ihop till ett färdigt läkemedel. En viktig del i den här metoden är att få fram kristaller av proteinerna, och med hjälp av röntgenstrålning få strukturell information om hur de ser ut. Därefter kan man söka efter de små molekylhämmarna och förbättra dem med målet att blockera virusets möjligheter att föröka sig.
Forskargruppen har därför påbörjat studier av dessa tre proteiner från SARS CoV-2.
Tillverkat kristaller av Nsp10
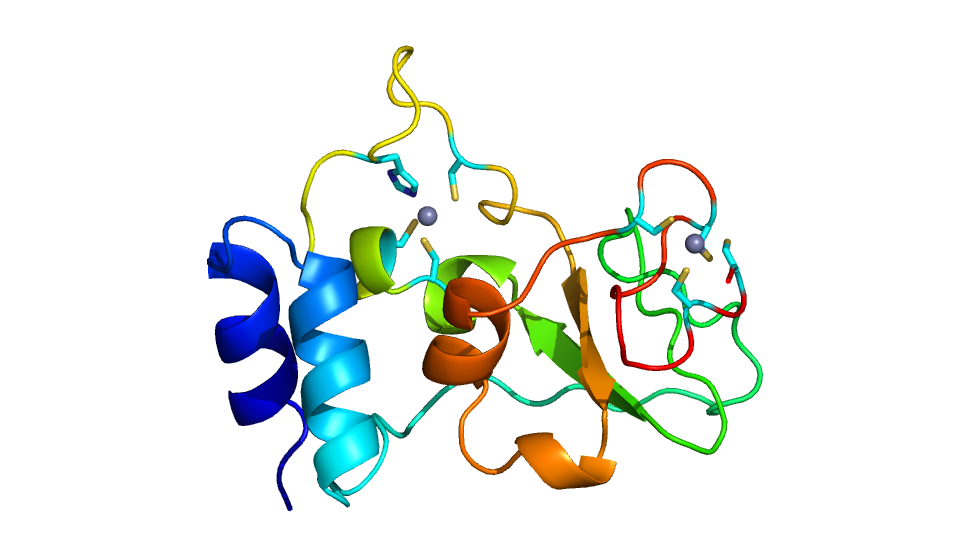
Just nu arbetar forskarna med att ta fram högupplösta modeller av kristallstrukturerna hos de tre proteinerna. Arbetet kommer att kompletteras med biofysiska och biokemiska karakteriseringsexperiment – som termisk stabilitet, interaktionsstudier proteinerna emellan och strukturanalyser med proteinerna i lösning med röntgen- och neutronspridning. Nyligen lyckades forskningsgruppen producera kristaller av ett av dessa proteiner, Nsp10 (se bild nedan).

Genom att snabbt få tillgång till strålröret BioMAX på MAX IV, samlades data in till cirka 2.6 Ångström, vilket är måttet på en upplösning där man kan få en detaljerad bild på molekylnivå.
Nu arbetar forskningsgruppen vidare med att få fram kristallstrukturerna även av proteinerna Nsp14 och Nsp16.
MAX IV-laboratoriet gör det osynliga synligt
MAX IV-laboratoriet är en nationell forskningsanläggning med Lunds universitet som värduniversitet. Forskare från hela världen kommer varje år till labbet och använder röntgenstrålarna i vetenskaplig forskning för att göra det osynliga synligt. De forskar inom bland annat fysik, kemi, geologi, ingenjörs- och materialvetenskap, strukturbiologi, medicin och nanoteknologi.
MAX IV-laboratoriet invigde i juni 2016 en ny anläggning vilken är en världens ljusstarkaste synkrotronljusanläggningar. Ungefär 270 personer är idag anställda vid MAX IV-laboratoriet.
I nuläget är 16 strålrör, de experimentstationer där forskningen bedrivs, finansierade och i olika stadier av färdigställande eller i drift och tar emot forskare för experiment. Totalt kommer cirka 25 strålrör att kunna installeras på de två lagringsringarna samt i förlängningen av linjäracceleratorn. Fullt utbyggt kommer MAX IV-laboratoriet att kunna ta emot upp mot 2 000 forskare per år.
Kontakt:
Professor Frank Kozielski, UCL School of Pharmacy, f.kozielski@ucl.ac.uk
Dr Wolfgang Knecht, Lunds universitet, wolfgang.knecht@biol.lu.se
Dr Zoë Fisher, ESS, zoe.fisher@ess.eu
Dr Ana Gonzalez, ana.gonzales@maxiv.lu.se
Dr Jie Nan, jie.nan@maxiv.lu.se